制御性T細胞研究の歴史について振り返る
制御性T細胞の根幹理論について再考する
私の20年にわたる免疫学研究の旅とTocky開発への道
免疫学の分野において、制御性T細胞(Treg)は自己免疫疾患の抑制や免疫寛容の維持において重要な役割を果たすとされています。しかし、このTregの存在とその機能に関する基礎的な理解には長い間議論が続いてきました。本ブログでは、1980年代から始まったTreg研究の歴史と、私自身の研究がTocky開発に至ることになる重要な要因について書きます。
抑制性T細胞理論の誕生
1980年代、Nishizukaは「抑制性T細胞(Suppressor T cells)」が出生後3日目以前には末梢に出現しないと予測しました。この予測は、d3胸腺摘出実験を通じて示唆されており、免疫学における数十年にわたる研究の基盤となりました。
Asanoらの1996年論文とその影響
1996年、坂口志文らが出版したAsanoらは、CD25⁺CD4⁺T細胞が出生後3日目以前には末梢に出現しないことを「確認」する論文を発表しました。この発見により、AsanoらはCD25⁺CD4⁺T細胞をNishizukaが予測した抑制性T細胞として位置付け、制御性T細胞(Treg)の重要な基盤を築きました。
この「画期的な」研究は、制御性T細胞(Treg)の主要な提唱者となったEthan Shevach博士を含む主流の免疫学者に熱狂的に受け入れられました。そしてShevachがTregの「伝道者」として世界にTregの研究を広める役割を担っていきます。
博士学生としての研究の開始
2002年、私は学内留学として京都大学皮膚科から京都大学再生医科学研究所の坂口志文教授の研究室で博士課程を開始しました。
私の研究テーマは、制御性T細胞(Treg)におけるGITRの役割についてでした。この研究を深めるために、坂口教授グループが発表した制御性T細胞に関する重要な論文の一つであるAsanoら、J Exp Med, 1996に注目しました。
Asanoらは、マウスの出生後3日目までにCD25陽性T細胞が現れないと報告しています。しかし、出生後4日目以降にはCD25陽性T細胞が急激に増加することも示しています。
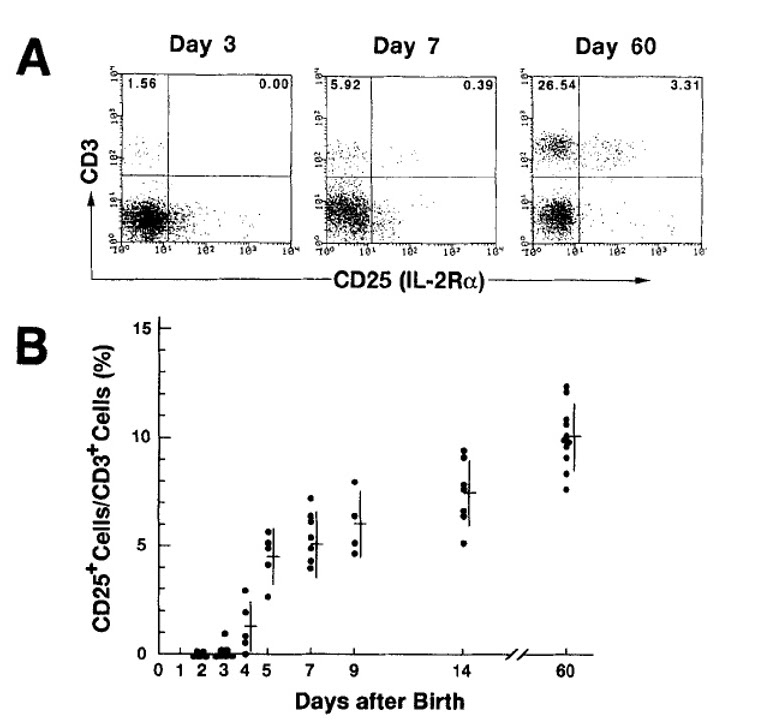
1990年代当時、CD25は活性化T細胞のマーカーとして広く使用されていました。この背景を理解することで、Asanoらの研究の意義が見えてきます。
つまり、CD25陽性T細胞が「胸腺で特別に発生し、自己免疫病を抑制する役割を持つ」という理論を支持する証拠として、Asanoらの新生仔期におけるCD25陽性T細胞の動態データは非常に重要でした。これが「制御性T細胞(Treg)が自己免疫病を抑制する特別なT細胞である」という現在も広く受け入れられている概念の確立につながりました。
再現性の確認
2003年、私は博士課程2年目に、Asanoらのデータを再確認する実験を行いました。結果は、Asanoらの論文とは異なり、出生後1日目の新生仔マウスの脾臓と胸腺には十分な数のCD25陽性T細胞が存在することが分かりました。2日目、3日目、7日目、1週間目、2週間目と、いずれの日齢でも大きな差は見られませんでした。
この結果は、Asanoらの1996年のTregに関する画期的な論文の主要な主張と明確に矛盾しています。しかし、何度追試しても私のデータは一貫して同じ結論を示しました。制御性T細胞の研究分野に何か決定的におかしい点があると確信しました。
また、私だけの発見ではありませんでした。バンデイラ研究グループによるDujardinら、PNAS, 2004のデータも、私が発見した結果と一致していました。
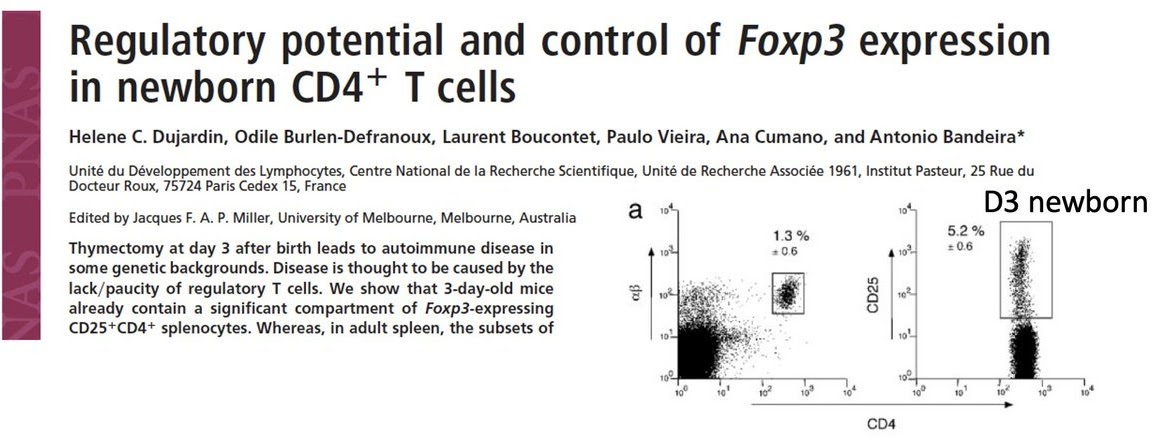
現状と課題
当時、私はAsanoらの1996年の研究成果に大きく依存している制御性T細胞理論が間もなく改訂されると信じていました。しかし、現実はそうではありませんでした。多くの教科書や総説論文では、新生児の制御性T細胞の動態に反する多くの証拠があるにもかかわらず、Asanoらの発見が紹介され続けていました。
この状況を受けて、2015年に私はAsanoらの論文の問題点について検証する論文を発表しました。これにより、Asanoらの「発見」を紹介する教科書や論文の数は減少しましたが、最近でも例えばSvoboda, Nature, 2022の制御性T細胞に関する記事などで、時代遅れとなったAsanoらの論文が紹介されています。

制御性T細胞研究の見直しと未来
この経験から、私は制御性T細胞分野で広く受け入れられている論文や理論を厳密に検証することの重要性を痛感しました。さまざまなアプローチを用いた研究により、Tregの理論と概念自体に大幅な見直しが必要であると確信しました。
この認識に基づき、制御性T細胞、ひいてはT細胞全体を新しい視点で研究するための新規研究方法の開発が必要であると理解しました。2009年には、ヒエラルキーから離れ、自分の研究分野を拡大し自由に研究するために、日本から英国へ移動することを決断しました。
この取り組みは、後にゲノミクスにおける多次元解析方法や正準対応解析(Canonical Correspondence Analysis, CCA)およびTockyの開発につながりました。
「制御性T細胞神話」と今後の展望
Asanoらの論文の問題は、免疫学の広範な分野の発展と研究を阻害する深刻な影響を及ぼしました。そこで、私はこれらの問題を検証し、過去の論文から得られる証拠を分析し、新たな視点を提案する論文を執筆・出版しました。
さらに、Asanoら、1996の論文に基づく「制御性T細胞がユニークで特別な細胞である」という固定観念が、多くの研究において研究デザインや結果の解釈を歪め、科学的真実から遠ざけている現状があります。
これは、1細胞分析やFoxp3に関する高度な解析など、最先端技術を用いた制御性T細胞研究においても同様です。間違ったデザインと解釈は、誤った結果を導くだけです。誤った論文を信じ続けることで、免疫学の発展が停滞しているのです。
したがって、「ユニークで特別な細胞」という固定観念を捨て、Foxp3および制御性T細胞を新しい枠組みで分析する研究方法の確立が急務です。
これからも、科学的真実に基づいた研究を推進し、免疫学の発展に貢献していきたいと考えています。 この問題意識が私たちのTocky論文第2弾である、Foxp3転写の時間動態解析につながります (Bending et al., 2018)。はEMBO Journalに掲載されたこの論文の内容については次の記事「制御性T細胞応用を目指した臨床試験の悲劇」で紹介します。
References
2018
- A temporally dynamic Foxp3 autoregulatory transcriptional circuit controls the effector Treg programmeThe EMBO journal, Sep 2018The second Tocky paper from the Ono lab uncovers the temporally dynamic regulation of Foxp3 transcription, offering new insights into T cell regulation.